Microbial vertical diversity in core sediments and its response to environmental factors near the hydrothermal field of the southern Okinawa Trough
-
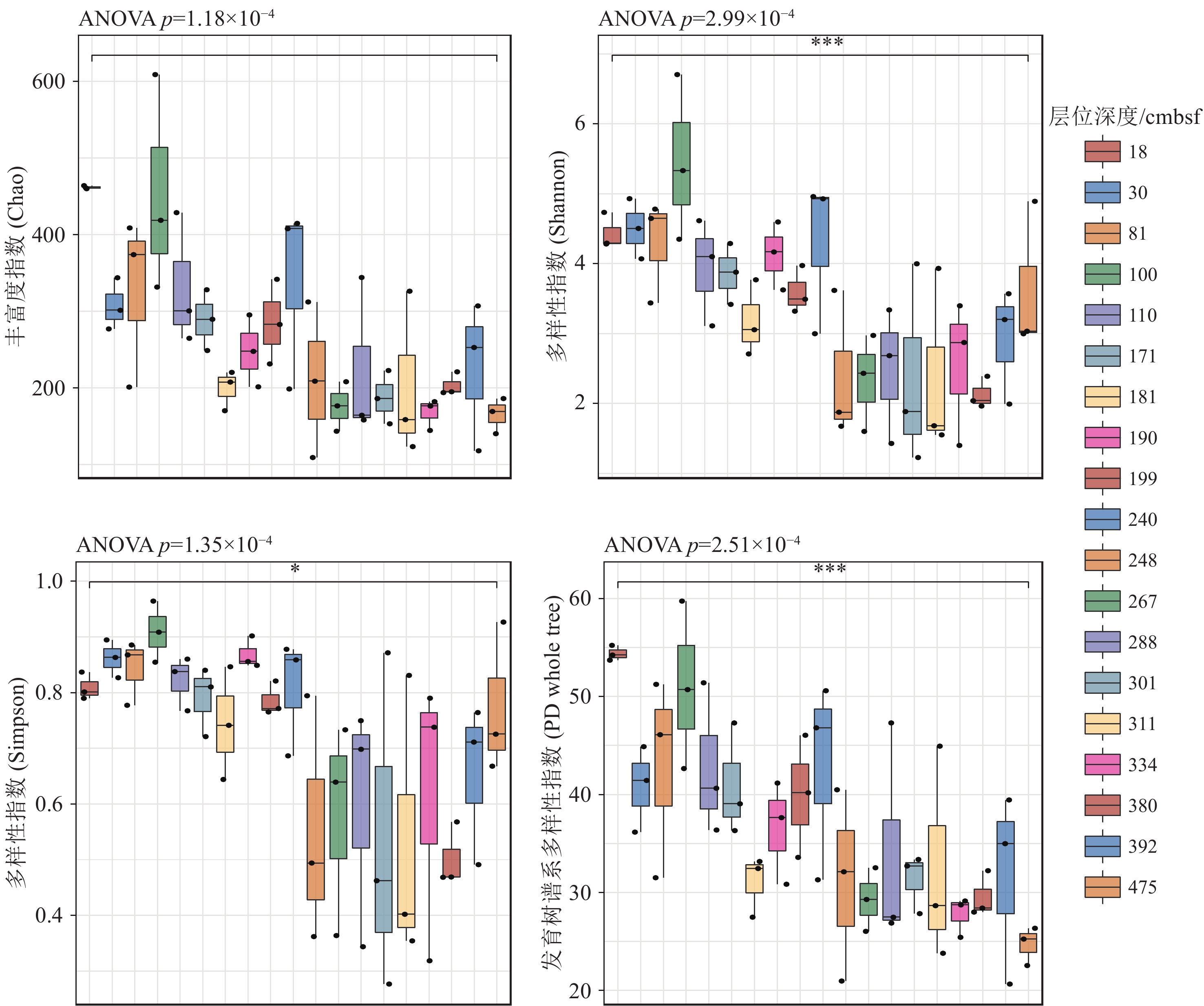
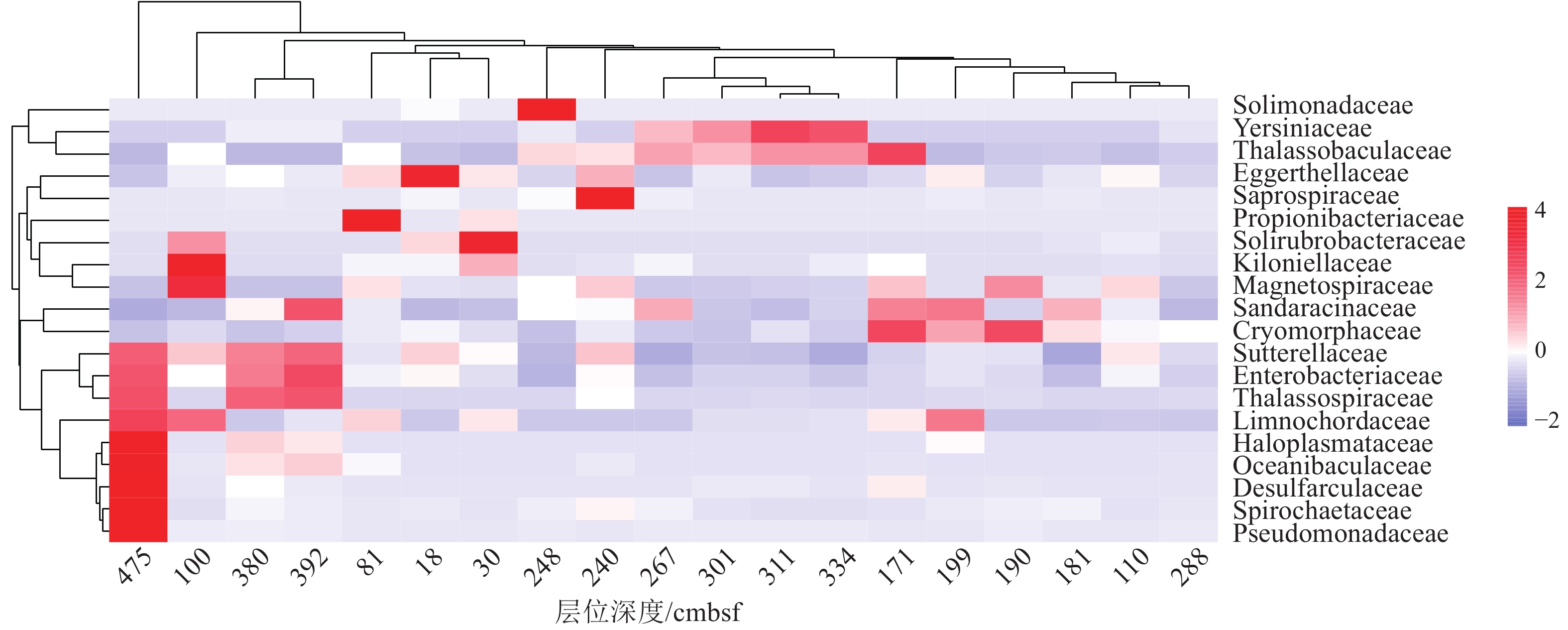
摘要: 近年来,海底热液环境中的微生物及其环境适应机制已经成为海洋科学研究的热点。目前,相关的研究主要集中在表层沉积物及微生物的水平分布多样性方面,而对柱状沉积物中微生物垂直分布多样性研究却很少。本文基于西太平洋冲绳海槽南部热液区附近S2站位的柱状沉积物样品,通过对其不同层位的样品进行分离培养和16S rRNA基因高通量测序,揭示了样品中可培养微生物和总体微生物的垂直群落分布特征,同时结合对样品主量元素、微量元素、碳氮含量等指标的评估和冗余分析等统计学方法,讨论了微生物群落结构及其对环境因子的响应。研究发现该位点的柱状沉积物有机质含量较为贫乏,存在两个富含Cu-Zn-Pb的层;各个层位的沉积物中微生物类群均以变形菌为主要类群,同时表层沉积物表现出更高的微生物多样性。此外研究还表明柱状沉积物中有机碳含量与其微生物的群落组成有着更为密切的关系。总之, 本研究的结果和获得的菌种资源为进一步深入研究海底热液环境中微生物参与元素地球化学循环的过程提供了一定的基础。Abstract: In recent years, microbe and its adaptation mechanism in submarine hydrothermal environment have become the focus of marine science research. At present, relevant researches focus on the horizontal distribution diversity of surface sediments and microorganisms, and only few researches on the vertical distribution diversity of microorganisms in columnar sediments. Based on the columnar sediment samples from the S2 station in the southern hydrothermal area of the Okinawa Trough in the western Pacific Ocean, we revealed the vertical community distribution characteristics of culturable microorganisms and the overall microorganisms in the samples through isolation, culture, and high-throughput sequencing of 16S rRNA gene from the samples at different levels. At the same time, the microbial community structure and its response to environmental factors were discussed by using statistical methods such as the evaluation of major elements, trace elements, carbon and nitrogen contents, and redundancy analysis. Results show that the organic matter content of the core sediment at this site is relatively poor, and Cu-Zn-Pb is rich in two layers; the microbial community of each layer is composed of mainly Proteobacteria, while the surface sediments exhibit higher microbial diversity. Meanwhile, it indicated a closer relationship between the organic carbon content of core sediments and the composition of their microbial communities. This study obtained bacterial strain resources and provided a basis for further research into microbial participation in the geochemical cycle of elements in submarine hydrothermal environment.
-
生物硅(BSi),又称生物蛋白石,主要由上层水体中的硅质浮游生物(如硅藻、放射虫、硅鞭藻等)死亡后的骨骼或细胞壁堆积而成[1-2],是海洋沉积物中重要的生源物质组成部分[3],其含量与表层水体中的生物量密切相关[4]。在上述硅质生物种群中,硅藻遍布于各类水体,为海洋提供了40%左右的初级生产力[5-6],是海洋BSi中最主要的组成部分[7-8]。因此,沉积物BSi的时空分布常作为古海洋初级生产力的变化指标[9]。由于生产力的波动与表层海水营养物质的供应量关系密切,因此可以将BSi沉积记录与可能导致海水营养盐供给变化的大尺度气候和海洋过程(如季风、洋流等)联系起来,作为重建过去海洋环境变化的重要工具[10-12]。
南海是西太平洋最大的边缘海,地理位置独特,其环境受东亚季风的强烈影响,是追溯冰期-间冰期旋回中东亚季风演变历史的热点区域之一[13-15]。泰国湾(Gulf of Thailand),旧称暹罗湾,是南海重要的组成部分,位于南海西南部的巽他陆架之上,处于太平洋及其附属海的最西端,是南海最大的海湾之一,为典型的半封闭型陆架海,其面积约35 000 km2。泰国湾属于热带季风气候,其风向和海流与亚洲季风关系密切[16-17]。在西南季风影响的5—10月,该海域沿岸流呈顺时针运动,而在受东北季风影响的11月至次年2月沿岸流则呈逆时针运动[18]。近年来有关研究表明,南海古生产力的变化与亚洲季风的周期性变化具有密切联系[10, 12, 19-22],然而这些研究主要集中在南海海盆和北部沿岸地区,有关南海西部和西南部,尤其是泰国湾的古生产力研究则少有涉及。因此本研究选取位于泰国湾北部沿岸海域的两根沉积物柱状样LCS05和WLE08,通过对柱样中BSi含量的分析,探讨近9 ka以来泰国湾古生产力的状况。
1. 材料与方法
1.1 样品采集
本研究所用的LSC05和WLE08沉积柱样,系由自然资源部第三海洋研究所和泰国自然资源与环境部矿产资源局合作于2017年5月12日在泰国湾尖竹汶海岸通过重力及回旋取样结合的方法取得。其中,LSC05柱样(12°21′29″N、101°58′36″E)长1.58 m,水深18 m;WLE08柱样(12°17′28″N、102°10′18″E)长1.98 m,水深13 m[17],具体采样位置见图1。在自然资源部第三海洋研究所海洋与海岸地质实验室以2 cm连续间隔对两根柱样进行分样。LSC05柱样共挑选27个样品进行BSi(Si)和有机碳(TOC)测试分析;WLE08柱样共挑选33个样品进行BSi(Si)和有机碳(TOC)测试分析。有机碳(TOC)分析在自然资源部第三海洋研究所完成;BSi分析工作在中国海洋大学海洋地球科学学院地球化学实验室完成。
1.2 生物硅分析
BSi测试采用钼酸铵分光光度法测定萃取物中溶解的二氧化硅[23]。对沉积物样品研磨烘干(65℃烘48 h,106℃下烘2 h),称取130~140 mg干样于50 mL塑料离心管中,加5 mL10% 的H2O2溶液,混匀后静置30 min。加5 mL 1∶9的HCL混匀,静置30 min。加20 mL去离子水,离心20 min,之后对样品进行干燥。干燥后的样品加40 mL 浓度为2 mol/L的 Na2CO3提取液,放置在85 ℃水浴中加热提取。之后每隔1 h取出离心,取125 µL上清液用于测定Si浓度,连续提取8 h。利用分光光度法测定提取液中的硅[24-25]。测得的提取液中的Si含量数据和对应的提取时间点作图,曲线直线部分的反向延长线与Y轴的交点为样品的BSi含量[23-24]。全过程标准偏差控制在5%以内。
1.3 AMS14C测年分析
根据粒度测试结果选取LSC05柱样的4个层位和WLE08柱样的3个层位中的贝壳或植物碎屑样品进行AMS14C测试。样品送美国BETA实验室完成,BETA实验室已对原始测年数据进行了日历年龄校正,海洋碳库效应校正由于附近没有已知点故选取全球海洋修正,具体测试结果见表1。本文年代采用日历年。
柱样名称 样品深度/cm BETA实验室编号 测年材料 AMS14C测量年代/(aBP±1σ) 日历年龄/cal. aBP LSC05 8 Beta-509448 贝壳 3090±30 2857 18 Beta-509449 贝壳 3210±30 3024 100~102 Beta-509450 植物碎屑 6880±30 7726 152~154 Beta-509451 植物碎屑 8480±30 9499 WLE08 12~14 Beta-509452 贝壳 350±30 − 96~98 Beta-509453 贝壳 1550±30 1109 184~186 Beta-509454 贝壳 1S820±30 1373 1.4 有机碳测试
有机碳(TOC)分析采用Vario Isotope cube-IsoPrime 100型(Elementar)元素分析-稳定同位素比值质谱联用仪(EA-IRMS)完成。前处理取一定量的沉积物样品,加入4 mol/dm3 的HCl至过量,反应24 h。用去离子水洗酸至中性,将样品置于烘箱内60 ℃烘干,恒重后称量,研磨成粉末,过60目的筛子,密封备用。用天平准确称取适量固体样品,放入锡箔杯中并紧密包裹成小球状,依次放进96孔板内测定。
2. 结果与讨论
2.1 年代地层
LSC05、WLE08柱样的AMS14C测年数据如表1,两根柱样的年龄与深度模型见图2、3。在沉积物样品测年基础上,通过线性插值,获得各样品所代表的年龄,进而建立柱状样的年代框架。沉积速率的计算是通过两个测年点的线性关系得到,通常不考虑沉积物的固结压实和沉降作用。对于柱样第一个测年点之上的沉积速率,选择通过前两个测年点间的沉积速率外推至上部样品。LSC05柱样的沉积物年龄为2.7~9.6 cal.kaBP,18 cm以上的平均沉积速率约为59.9 cm/ka,18~100 cm的平均速率为17.4 cm/ka,100~158 cm的平均速率为29.3 cm/ka。WLE08柱样的年代框架已建立,建立方法与LSC05柱样相同。其沉积物年龄为1.1~1.4 cal.kaBP,整根柱子的沉积速率均为333.3 cm/ka [17]。LSC05孔提供了9 ka以来平均分辨率达约2.3 ka的沉积记录,WLE08孔提供了1.3 ka以来平均分辨率达约0.3 ka的高分辨率的沉积记录。
LSC05与WLE08柱样均从表层(0 cm)开始向下取样,但两根柱样均出现了顶部近代沉积物缺失的情况。根据WLE08柱样的210Pb测试结果显,示其活度随深度增加并没有呈现指数衰减趋势[27],说明 WLE08 孔的210Pb 活度值为本底值,缺少现代沉积物留存。有研究表明汇入该区域内的尖竹汶河与Welu河流量较小,入海泥沙量也相对较小[27],而其所在海域底层流流速均大于泥沙颗粒启动流速[28],在此沉积动力环境下,该区域很难大量接收现代河流沉积,并导致现代沉积物缺失。
2.2 有机碳含量及其古海洋学意义
除了BSi以外,直观反映了海表有机质丰度来体现海表初级生产力的有机碳含量(TOC)被视为衡量海表生产力的替代指标[29-30]。LSC05和WLE08柱样TOC含量变化范围分别为0.03%~1.60%和1.83%~4.10%,平均值分别为0.90%和2.68%(图4B)。TOC含量在9~6.5 cal.kaBP期间逐渐增加,在6.5~3 cal.kaBP期间含量整体较低,变化较小,较为稳定,在1.4~0.84 cal.kaBP期间含量较高。
![图 4 LSC05、WLE08柱样BSi含量、TOC含量、C/N比值与其他气候、环境变化序列对比]() 图 4 LSC05、WLE08柱样BSi含量、TOC含量、C/N比值与其他气候、环境变化序列对比粉红色条带代表BSi含量高值带,粉蓝色条带代表BSi含量低值带。Figure 4. Comparison among biogenic silica content, TOC content, C/N in LSC05, and WLE08 with other climate and environmental variation sequencesRedish bands represent the high value band of BSi content and the bluish bands represent the low value band of BSi content.
图 4 LSC05、WLE08柱样BSi含量、TOC含量、C/N比值与其他气候、环境变化序列对比粉红色条带代表BSi含量高值带,粉蓝色条带代表BSi含量低值带。Figure 4. Comparison among biogenic silica content, TOC content, C/N in LSC05, and WLE08 with other climate and environmental variation sequencesRedish bands represent the high value band of BSi content and the bluish bands represent the low value band of BSi content.在全球变暖的背景下,泰国湾的有机碳的输送、扩散和埋藏与热带季风气候控制的降水等条件密切相关[31]。研究区的TOC自6.5 cal.kaBP以来的变化趋势与王承涛等[17]研究得到的泰国湾中晚全新世以来沉积物的敏感粒级变化趋势吻合度较高,指示了研究区的西南夏季风在6.5~3 cal.kaBP处于较弱的稳定期,在1.4 cal.kaBP以来有所增强。在6.5 cal.kaBP以前的时期,本研究TOC含量较低,与中晚全新世以来沉积物的敏感粒级的研究结果差异较大,可能是由于王承涛等[17]研究的WLE12柱样位于河口内,但LSC05柱样距河口位置明显更远,因此LSC05接收到的来自河流携带的有机物汇入较少。
2.3 生物硅含量及其影响因素
LSC05和WLE08柱状沉积物中BSi含量如图4A所示。LSC05和WLE08柱样BSi含量变化范围分别为0.41%~1.56%和0.60%~1.52%,平均值分别为0.88%和1.11%。结合研究区周边与南海部分海域的BSi研究成果(表2)可以发现,研究区属于南海中的低值海区,与南海南部(巽他陆架东部)和南海北部陆坡(珠江口外)水深小于200 m的陆架浅水区沉积物BSi含量[19, 32]较为接近。
表 2 南海部分海域BSi含量对比Table 2. Comparison of BSi content in the South China Sea研究海域 沉积物性质 水深/m BSi含量占比/% 参考文献 范围 平均值 南海西部泰国湾(本文) LSC05柱样 15~20 0.41~1.56 0.88 — WLE08柱样 10 0.60~1.52 1.11 南海北部陆坡 ODP1144钻孔 2 037 — 1.50( 1050 ~900 ka)[42-43] 3.80(900 ka后) 南海北部 表层沉积物 <200 — 1.59 [19] >200 — 2.06 南海中部 ODP1143钻孔 2 722 1.31~3.38 - [22, 43] 南海南部 表层沉积物 <200 0.37~1.86 — [32] >1 000 3.39~9.00 — 东印度洋爪哇岛以南 CJ01-185柱样 1 538 — 1.41 [3] 沉积物中BSi含量的最主要的影响因素是硅质骨骼或细胞壁的供给量和溶解作用[33],两者的动态平衡关系主导了BSi的总体分布[34],再加上不同沉积物物质来源以及物质稀释的影响,共同决定了BSi最终的分布格局。LSC05和WLE08柱样采样站位水文环境条件与其他南海相关研究有所不同,季风的盛行导致南海存在较多的上升流区[35-37]。上涌的下层水体将营养物质携带至海表,为海表硅藻等硅质生物勃发提供了良好的生境,因此上升流区BSi含量明显较高。除本研究区以外,表2中涉及的其他站位均靠近南海上升流区[38-39],但有研究表明泰国湾区域无上升流发育[36],这就导致了泰国湾硅质骨骼与壳体供给量低于其他有上升流的南海海域,因此BSi含量偏低。
碳氮比值(C/N)是古生产力研究中重要的参考性指标 ,可以用来反映沉积物的来源[40]。海洋自身有机物的C/N一般为5~8[31, 41],陆源有机物的C/N则大于12[31]。研究区9 ka以来,LSC05和WLE08柱样的C/N比值偏高,平均值为14.81,较多时期的C/N比值高于12(图4C, 8.6~8.8 cal.kaBP和9.0~9.4 cal.kaBP数据缺失),说明研究区中陆源有机质输入量较高。LSC05与WLE08两根柱样的采样位置均位于近岸区域,水深为10~20 m,其沉积物物源受到海洋与陆地的双重影响。末次冰消期以来,泰国湾的沉积物均来自于中南半岛[44] ,较多陆源沉积物的输入稀释了BSi含量。
由此可见,泰国湾低BSi含量这一特征与BSi的来源、堆积过程及其保存环境密切相关,低硅质骨骼与壳体供给量和陆源物质的稀释共同作用形成了该BSi低含量海域。
2.4 生物硅指示的古生产力演化过程
沉积物中的BSi与有机质关系密切[45],虽然只有约 3%的BSi能长期存在于海底沉积物中,但是相对于生源有机碳,BSi的埋藏效率明显更高,更能保存上层水体的生产力及环境信息[46]。因此BSi常作为指示生产力的有效替代性指标,可以用来指示表层水体生产力的演变。
研究区BSi含量最高值出现于7.5 cal.kaBP,最低值出现于2.8 cal.kaBP,最高值为最低值的3.8倍。柱样中的BSi含量共有4个高值带,分别是8.1~7.5、4.8、3.4和1.0 cal.kaBP左右。同时有3个低值带,分别是8.2、7和3 cal.kaBP左右。
海洋初级生产力是各种物理、化学和生物因素的综合效应的表现[47]。东亚冬季风对于南海的生产力起着重要的作用[4,15,18,22,48-49]。亚洲季风的变化引起泰国湾降水、水体营养浓度、叶绿素含量、浮游植物丰度等环境变化[31, 48- 50]。若亚洲夏季风势力强盛,带来大量的降雨,使地表河流径流量增加,从而携带更多营养物质汇入海洋[10],BSi含量则随之增加,这就反映了海表初级生产力提高。
1.4~0.8 cal.kaBP期间,BSi含量为0.60%~1.52%(平均含量1.11%),属于本研究中偏高的一段时期。此时正处于东亚夏季风偏强的中世纪暖期时期,这说明该时期内的海表初级生产力较高可能是由于夏季风势力较强所致。8.5~8.2、7.1~6.9 cal. ka BP两段时间内,BSi含量较低,这可能是由于此时南亚夏季风势力较弱导致的。8.2 cal.kaBP时,BSi含量快速下降并近乎到达最低值。此时夏季风也处于该时期内最低值,表明夏季风存在一次较大的衰弱期,响应了8.2 cal.kaBP全球范围的气候突变事件[17,51],指示了此段时间内由于夏季风的衰减致使研究区海表初级生产力降低。
对比研究区柱样BSi含量曲线和南海区域温度异常值曲线[52](图4A、D)可以发现,温度变化与BSi含量变化也有相近的变化趋势,但BSi含量的变化略滞后于南海区域温度异常的变化。这反映出BSi作为古生产力的替代指标与古气候在一定程度上具有对应性[4]。但我们发现8.2 cal.kaBP时期冷事件之前的曲线呈现相反的变化趋势,南海温度异常增加,BSi含量反而降低,这反映了由于夏季风过度强盛而引起的异常暖事件反而可能会抑制硅质生物的生长,导致海表初级生产力降低[10]。
将BSi含量与格陵兰冰芯[53]、华南董歌洞石笋[54]以及阿曼Qunf岩洞石笋记录[55](图4F、G、H)对比,可以发现泰国湾9 ka以来有部分时期能够吻合。3~2.7 cal.kaBP时期,BSi含量出现明显低值带,此时期董歌洞石笋与阿曼Qunf岩洞石笋记录均出现了对应的低值区,可能指示了一次冷事件的出现,这与4~2 cal.kaBP时期出现的热带海域“斜氏普林虫低值事件”对应[56-57]。8.2 cal.kaBP变冷事件时期[51],格陵兰冰芯、董歌洞石笋以及阿曼Qunf岩洞石笋记录同样能找到相应的低值记录,格陵兰冰芯甚至达到最低值。以上对应事件说明研究区古生产力的变化趋势与全球尺度的环境变化具有相关性,指示了泰国湾古环境变化对全球环境变化有一定程度的响应。
3. 结论
(1) LSC05和WLE08柱样中BSi含量分别为0.41%~1.56%和0.60%~1.52%,为低值海域,最高值和最低值分别出现于7.5、2.8 cal.kaBP时期。区域内无上升流导致的低硅质骨骼和低壳体供给量以及陆源物质输入的稀释是BSi含量低的主要原因。
(2)本研究柱样中共出现了4个BSi含量高值带与3个BSi含量低值带。TOC含量自9 cal.kaBP以来呈现逐渐增加-平稳较低-继续增加的趋势。将 9 ka以来研究区生物硅含量曲线与南海气候曲线进行对比发现,生物硅含量与南海气温异常曲线和南海夏季风替代指标值期有一定的对应性,但BSi曲线略滞后于南海气温异常曲线,且1.4~0.84 cal.kaBP这一海表生产力高值期正处于东亚夏季风偏强的中世纪暖期时期,指示了高生物硅含量对应的海表高初级生产力时期可能是由于夏季风阶段性势力较强导致。但8.2 cal.kaBP冷事件之前的南海异常高温却对应了生物硅含量低值区,这可能反映了夏季风过强引起的过度高温反而不利于海表初级生产力的提高。
(3)研究区生物硅含量与格陵兰冰芯、董歌洞石笋以及阿曼Qunf岩洞石笋记录有较好的对应性,BSi含量低值带与热带海域“斜氏普林虫低值事件”和8.2 cal.kaBP冷事件对应,表明研究区古生产力的变化趋势与全球尺度的环境变化具有明显的相关性,这指示了泰国湾古环境变化对全球环境变化的响应。
致谢:感谢所有在航次调查期间帮助采样和采集数据的中泰合作项目组成员。
-
图 2 基于16S rRNA基因序列构建的可培养细菌的系统发育树
分支1中包含的菌株有18A01、18E05、18S01、81A02、81E07-1、199A01、301A01、334E05、334M01、334S02、334S04、392A01、392E02、475E05和Alcanivorax xenomutans JC109T (HE601937)。
Figure 2. Phylogenetic tree of cultivable bacteria isolated from hydrothermal field sediment core in the southern Okinawa Trough based on the 16S rRNA gene sequences using the maximum-likelihood algorithm
GenBank accession numbers are shown in parentheses. Bar, 0.1 substitutions per nucleotide position. Branch 1 represented 18A01, 18E05, 18S01, 81A02, 81E07-1, 199A01, 301A01, 334E05, 334M01, 334S02, 334S04, 392A01, 392E02, 475E05, and Alcanivorax xenomutans JC109T (HE601937).
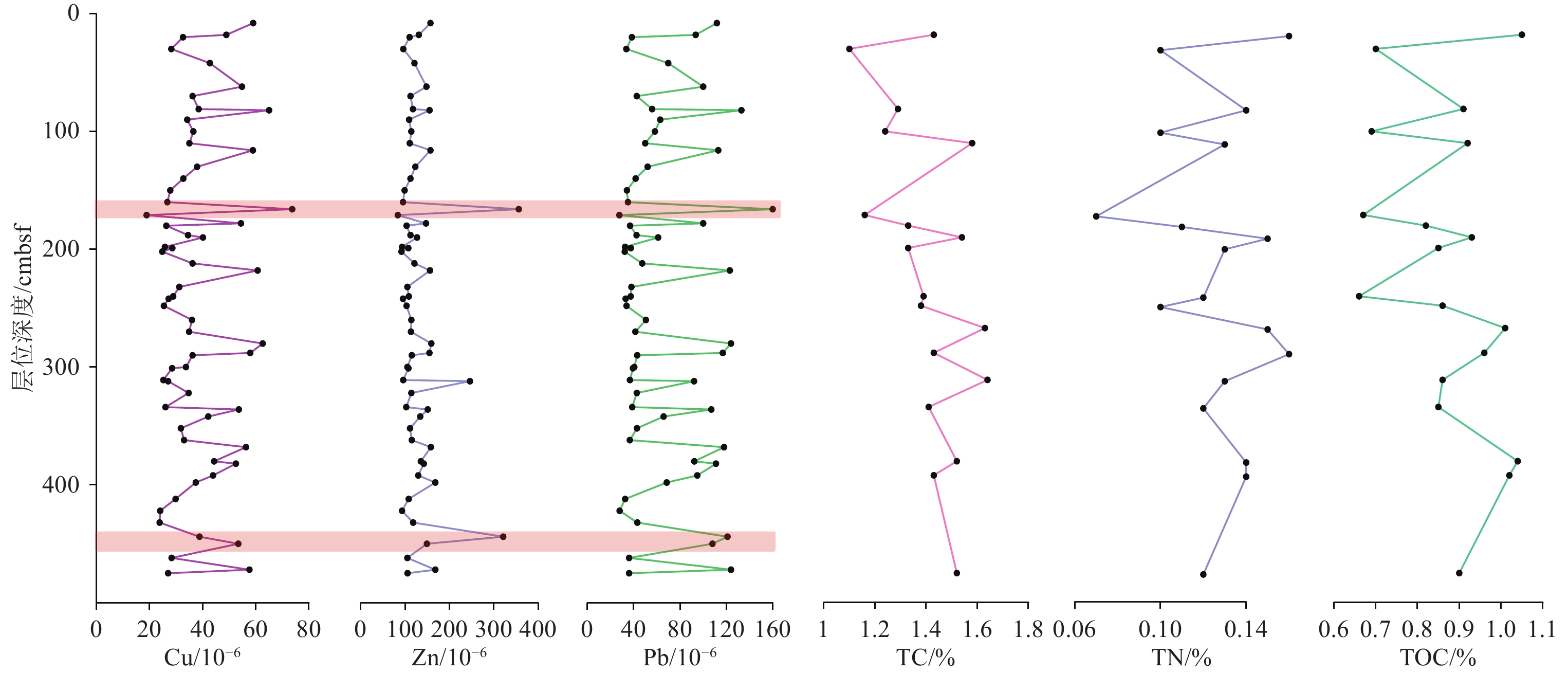
表 1 本研究使用样品的地球化学组成
Table 1 Geochemical compositions of the samples used in this study
样品深
度/cmbsf18 30 81 100 110 171 181 190 199 240 248 267 288 301 311 334 380 392 475 Na2O/% 2.03 1.98 1.81 1.9 1.86 1.98 1.8 1.84 1.84 1.79 1.79 1.78 1.95 1.89 1.96 1.83 1.89 2.02 1.88 MgO/% 2.64 2.3 2.49 2.5 2.59 2.03 2.43 2.65 2.58 2.4 2.38 2.41 2.71 2.58 2.48 2.39 2.54 2.57 2.49 Al2O3/% 16.32 15.39 16.33 15.77 15.23 14.25 16.11 16.65 16.31 16.37 16.2 16.41 16.13 15.92 15.31 15.98 15.96 15.9 16.37 SiO2/% 58.88 60.43 59.27 59.7 59.17 62.59 59.43 58.05 57.99 59.26 59.53 59.27 59.12 58.48 58.99 59.51 59.62 58.96 58.55 Fe2O3/% 6.43 5.85 6.43 6.03 5.98 5.09 6.21 6.41 6.43 6.31 6.18 6.23 6.32 6.23 5.8 6.14 6.26 6.24 6.26 K2O/% 3.26 2.97 3.27 3.11 3.09 2.76 3.26 3.46 3.35 3.29 3.23 3.26 3.28 3.23 3.04 3.21 3.24 3.17 3.28 CaO/% 2.21 2.67 2.4 2.66 3.52 2.92 2.6 2.66 2.92 2.55 2.6 2.49 2.41 3.13 3.46 2.68 2.52 2.62 2.75 P/10−6 706.1 714.3 684.3 674 632.5 623.6 631.4 607.2 635.4 642 649.2 689.1 646.6 602.5 653.1 620.1 623 899 618.5 S/10−6 761.1 859.7 649.3 841 804.9 956.8 567.1 580.2 657.2 517.6 559 569.4 620.3 650.5 782.9 556.5 734.1 660.4 661.3 Cl/10−6 6037.2 6000 3860.6 4979.2 4420.3 5220.8 4230.2 5057 5537.6 3815.5 4061.6 4257.9 5084 5536.1 6376.4 4519.6 4159.3 6186.9 5395 Ti/10−6 4742.1 4697.5 4834.1 4714.8 4530.7 4377.6 4777.1 4603 4734.4 4868.5 4826 4845.9 4641.3 4639.4 4567 4743.7 4663.3 4562.8 4811.9 V/10−6 131.8 110 128.4 124.9 122.7 100.9 122.7 129.6 128.4 118.9 119.6 117.4 139.2 127.3 117 119.8 131.9 129.2 125 Cr/10−6 99.5 84.6 94.9 88.2 88.8 76.1 90.1 100.1 95.2 91.6 90.8 90 97.6 92.6 85.8 88.6 94.6 89.8 93.4 Mn/10−6 478.4 505.3 527.1 463 496.2 404.1 507 475 493.8 513.2 510.5 542.4 489.8 503.1 492.1 509.3 458.6 623.8 529.6 Co/10−6 16.5 16.6 18.6 16 21.8 13.9 16.6 16.7 16.1 15.6 15.6 17.7 17.7 17.5 16 17 16.7 16 16.8 Ni/10−6 40.3 35.8 40.8 37.2 36.9 30.8 37.5 39.9 38.5 38.5 37.3 37.5 39.4 39.5 35.9 36.9 38.8 37.3 39 Cu/10−6 48.9 28.2 38.5 36.5 35 18.9 26.3 40.1 28.6 28.9 25.4 26.5 57.9 28.5 25.2 26 44.3 43.9 27 Zn/10−6 130.8 95.9 117.6 113.9 110.4 83.4 103.3 126.8 107.2 108.3 103.2 105.5 154.7 107.4 95.7 102.5 135.4 129.5 105.4 Ga/10−6 21.2 19.7 22.5 20.2 20.9 18.3 21 22.9 21.6 22.8 20.9 21.6 21.3 21.7 19 21.5 22.2 20.7 21.7 As/10−6 17 15.5 19.4 12.3 10.5 8.3 12.2 14.8 12.5 12.6 12.2 13.2 17.3 11.6 9.2 11.5 14 14.7 11.7 Rb/10−6 150.6 135.9 153.3 141.8 140.8 122.5 152.7 163.4 158 157.3 151.8 152.6 150.5 151.1 137.3 150.6 149.7 144.6 154 Sr/10−6 144 149 149 149 170 149 149 155 159 151 152 147 151 162 167 152 151 155 155 Zr/10−6 194 208 190 203 189 236 196 170 177 192 198 196 184 179 200 194 189 191 187 Nb/10−6 18.3 21.6 18 17.3 17.7 17 18.8 17.7 20.4 18.5 19 18.7 17.8 19 18.9 18.5 21.1 17.8 18.2 U/10−6 3.7 3.8 3.5 3.5 3.9 2.9 3.8 3.9 4.2 4.1 3.8 3.8 3.8 3.8 3.4 3.8 3.9 3.6 3.6 Mo/10−6 1.2 1.5 1.3 1.1 1.3 1 1.4 1.5 1.6 1.3 1.3 1.3 1.4 1.4 1.3 1.3 1.7 1.2 1.3 Sn/10−6 10.1 9.7 8.3 7.3 10.6 8 12.2 14.6 13.6 9.5 11.1 14.2 10.9 14.4 15.2 10.8 18.7 13.6 10.9 Sb/10−6 15.2 9.6 9.3 7.1 10 11.2 13.4 18.3 13.8 7.7 12.3 15.7 11.8 20.6 17.2 13 24.5 15 14.9 Ba/10−6 524 474 521 493 507 430 517 549 515 509 500 497 562 504 487 503 537 518 500 Hf/10−6 5.5 5.9 5.4 6 5.5 6.7 5.5 5.1 5.1 5.7 5.9 5.5 5.3 5.4 5.8 5.5 5.2 5.6 5.3 W/10−6 2.9 7.3 5.1 12.1 9.1 12.9 4.4 2.4 6.3 4.3 4.2 6.9 11.6 3.2 7.6 4.3 4.4 6.6 4.6 Pb/10−6 93.5 33.6 55.9 58.3 49.9 27.6 36.8 61.1 37.5 37.4 33.8 34.5 116.9 39.3 36.7 38.7 92.3 94.9 36 Bi/10−6 0.5 0.2 1 1.3 1.4 2.4 1.2 1.6 0.3 1.3 1.5 0 0 0 0.7 1 0.6 1.3 0 Th/10−6 15.2 15 13.8 13.9 12.8 10.6 16.1 14 16.1 14.7 14.6 15.5 14.7 16.4 13.1 14.8 15.1 14.7 14.5 Ce/10−6 76.5 54.2 62.8 73.7 71.8 74.8 65.5 68.4 78.7 72.8 66.4 69.4 66.8 68.4 72.9 65.1 68 65.7 84.4 Nd/10−6 27.8 38.4 34 29.1 32.8 29.3 35.1 31 26.9 26.8 33.6 30.8 35.7 31.3 31.8 28 27.5 25.2 29.5 Y/10−6 26.8 27.4 26.8 27 25.5 26.1 26.8 24.4 26 26.2 27.7 27.2 25.8 25.8 26.1 26.7 25.4 25.6 27 La/10−6 44.7 36.3 44.3 35.3 36.4 40.3 41.2 36.4 44.6 44.8 39.8 42 37.7 38.1 33.8 40.8 38.2 44.6 43.8 Sc/10−6 13.9 15 16.6 14.7 15.5 10.6 16 15.6 16.1 13.6 12.9 17.5 13.4 14.3 12.8 15.9 15.1 15.4 14.3 TN/% 0.16 0.1 0.14 0.1 0.13 0.07 0.11 0.15 0.13 0.12 0.1 0.15 0.16 0.12 0.13 0.12 0.14 0.14 0.12 TC/% 1.43 1.1 1.29 1.24 1.58 1.16 1.33 1.54 1.33 1.39 1.38 1.63 1.43 1.62 1.64 1.41 1.52 1.43 1.52 TOC/% 1.05 0.7 0.91 0.69 0.92 0.67 0.82 0.93 0.85 0.66 0.86 1.01 0.96 0.98 0.86 0.85 1.04 1.02 0.9 表 2 基于16S rRNA基因序列的菌株分类鉴定信息
Table 2 Isolation and identification of strains based on 16S rRNA gene sequence analysis
菌株号 亲缘菌株 16S rRNA
基因相似性/%层位深度
/cmbsf16S rRNA基因
序列登录号18A01 Alcanivorax xenomutans JC109T 99.22 18 OQ186762 18E01 Bacillus tequilensis KCTC 13622T 99.79 18 OQ186763 18E02 Pelagibacterium halotolerans B2T 99.78 18 OQ186764 18E03 Bacillus altitudinis 41KF2bT 99.93 18 OQ186765 18E04 Pseudonocardia carboxydivorans Y8T 99.93 18 OQ186766 18E05 Alcanivorax xenomutans JC109T 100 18 OQ186767 18M01 Alcanivorax venustensis ISO4T 100 18 OQ186768 18S01 Alcanivorax xenomutans JC109T 100 18 OQ186769 81A02 Alcanivorax xenomutans JC109T 99.93 81 OQ186770 81E01 Rossellomorea aquimaris TF-12T 99.21 81 OQ186771 81E02 Sutcliffiella halmapala DSM 8723T 99.14 81 OQ186772 81E03 Rossellomorea aquimaris TF-12T 99.29 81 OQ186773 81E05 Fictibacillus arsenicus Con a/3T 99.71 81 OQ186774 81E06 Bacillus safensis subsp. safensis FO-36bT 99.86 81 OQ186775 81E07-1 Alcanivorax xenomutans JC109T 99.93 81 OQ186777 81E07-2 Staphylococcus pseudoxylosus S04009T 99.86 81 OQ186778 81E08 Mesobacillus thioparans BMP-1T 99.71 81 OQ186779 81E09 Mesorhizobium sediminum YIM M12096T 99.77 81 OQ186780 110E01 Halomonas titanicae BH1T 99.44 110 OQ186781 110E02 Metabacillus idriensis SMC 4352-2T 99.93 110 OQ186782 110E03 Pseudonocardia carboxydivorans Y8T 100 110 OQ186783 110E04 Mesorhizobium sediminum YIM M12096T 99.77 110 OQ186784 110E05 Pelagibacterium halotolerans B2T 99.78 110 OQ186785 110E06 Sutcliffiella horikoshii DSM 8719T 99.29 110 OQ186786 110E07 Virgibacillus halodenitrificans DSM 10037T 99.93 110 OQ186787 181E01 Halomonas titanicae BH1T 100 181 OQ186788 181E02 Rossellomorea aquimaris TF-12T 99.36 181 OQ186789 181E03 Pseudonocardia carboxydivorans Y8T 99.79 181 OQ186790 181E04 Nitratireductor aquibiodomus JCM 21793T 99.08 181 OQ186791 181M01 Halomonas titanicae BH1T 100 181 OQ186792 181S01 Martelella mediterranea DSM 17316T 99.63 181 OQ186793 199A01 Alcanivorax xenomutans JC109T 100 199 OQ186794 199E01 Nitratireductor aquibiodomus JCM 21793T 99.17 199 OQ186795 199E02 Mesorhizobium sediminum YIM M12096T 99.77 199 OQ186796 199E03 Pseudonocardia carboxydivorans Y8T 99.78 199 OQ186797 199M01 Halomonas titanicae BH1T 100 199 OQ186798 248E02 Paenisporosarcina quisquiliarum SK 55T 99.38 248 OQ186799 248E04 Nitratireductor aquibiodomus JCM 21793T 99.03 248 OQ186800 248E05 Citromicrobium bathyomarinum JF-1T 99.7 248 OQ186801 301A01 Alcanivorax xenomutans JC109T 99.93 301 OQ186802 301E02 Paenisporosarcina macmurdoensis CMS 21wT 99.31 301 OQ186803 301E04 Paenisporosarcina macmurdoensis CMS 21wT 99.72 301 OQ186804 301E05 Pelagerythrobacter marinus H32T 99.93 301 OQ186805 301E06 Nitratireductor aquibiodomus JCM 21793T 98.94 301 OQ186806 334E03 Paenisporosarcina macmurdoensis CMS 21wT 99.15 334 OQ186807 334E04 Paenisporosarcina macmurdoensis CMS 21wT 99.14 334 OQ186808 334E05 Alcanivorax xenomutans JC109T 100 334 OQ186809 334M01 Alcanivorax xenomutans JC109T 99.93 334 OQ186810 334S02 Alcanivorax xenomutans JC109T 100 334 OQ186811 334S03 Muricauda ruestringensis DSM 13258T 98.48 334 OQ186812 334S04 Alcanivorax xenomutans JC109T 100 334 OQ186813 392A01 Alcanivorax xenomutans JC109T 99.93 392 OQ186814 392E01 Paenisporosarcina quisquiliarum SK 55T 99.71 392 OQ186815 392E02 Alcanivorax xenomutans JC109T 100 392 OQ186816 392E03 Pelagerythrobacter marinus H32T 100 392 OQ186817 392E04 Pelagibacterium nitratireducens JLT2005T 99.77 392 OQ186818 392E05 Paracoccus marcusii DSM 11574T 99.77 392 OQ186819 392E06 Paenisporosarcina quisquiliarum SK 55T 99.79 392 OQ186820 475E01 Alkalihalobacillus hwajinpoensis SW-72T 99.65 475 OQ186821 475E02 Thalassospira xiamenensis M-5T 99.65 475 OQ186822 475E03 Qipengyuania vulgaris 022 2-10T 99.85 475 OQ186823 475E04 Pelagibacterium nitratireducens JLT2005T 99.7 475 OQ186824 475S01 Alcanivorax xenomutans JC109T 99.93 475 OQ186825 475M01 Thalassospira xiamenensis M-5T 99.85 475 OQ186826 -
[1] 王风平, 周悦恒, 张新旭, 等. 深海微生物多样性[J]. 生物多样性, 2013, 21(4): 446-456 WANG Fengping, ZHOU Yueheng, ZHANG Xinxu, et al. Biodiversity of deep-sea microorganisms[J]. Biodiversity Science, 2013, 21(4): 446-456.
[2] 李学恭, 徐俊, 肖湘. 深海微生物高压适应与生物地球化学循环[J]. 微生物学通报, 2013, 40(1): 59-70 LI Xuegong, XU Jun, XIAO Xiang. High pressure adaptation of deep-sea microorganisms and biogeochemical cycles[J]. Microbiology China, 2013, 40(1): 59-70.
[3] Takai K, Nakagawa S, Nunoura T. Comparative investigation of microbial communities associated with hydrothermal activities in the Okinawa Trough[M]//Ishibashi J I, Okino K, Sunamura M. Subseafloor Biosphere Linked to Hydrothermal Systems. Tokyo: Springer, 2015: 421-435.
[4] Takai K, Nakamura K. Compositional, physiological and metabolic variability in microbial communities associated with geochemically diverse, deep-sea hydrothermal vent fluids[M]//Barton L L, Mandl M, Loy A. Geomicrobiology: Molecular and Environmental Perspective. Dordrecht: Springer, 2010: 251-283.
[5] Nakamura K, Takai K. Theoretical constraints of physical and chemical properties of hydrothermal fluids on variations in chemolithotrophic microbial communities in seafloor hydrothermal systems[J]. Progress in Earth and Planetary Science, 2014, 1(1): 5. doi: 10.1186/2197-4284-1-5
[6] Nakagawa S, Takai K. Deep-sea vent chemoautotrophs: diversity, biochemistry and ecological significance[J]. FEMS Microbiology Ecology, 2008, 65(1): 1-14. doi: 10.1111/j.1574-6941.2008.00502.x
[7] Takai K, Nakamura K. Archaeal diversity and community development in deep-sea hydrothermal vents[J]. Current Opinion in Microbiology, 2011, 14(3): 282-291. doi: 10.1016/j.mib.2011.04.013
[8] Guan L, Cho K H, Lee J H. Analysis of the cultivable bacterial community in jeotgal, a Korean salted and fermented seafood, and identification of its dominant bacteria[J]. Food Microbiology, 2011, 28(1): 101-113. doi: 10.1016/j.fm.2010.09.001
[9] Thornburg C C, Zabriskie T M, McPhail K L. Deep-sea hydrothermal vents: potential hot spots for natural products discovery?[J]. Journal of Natural Products, 2010, 73(3): 489-499. doi: 10.1021/np900662k
[10] Wang S S, Jiang L J, Hu Q T, et al. Elemental sulfur reduction by a deep-sea hydrothermal vent Campylobacterium Sulfurimonas sp. NW10[J]. Environmental Microbiology, 2021, 23(2): 965-979. doi: 10.1111/1462-2920.15247
[11] 杜瑞, 于敏, 程景广, 等. 冲绳海槽热液区可培养硫氧化细菌多样性及其硫氧化特性[J]. 微生物学报, 2019, 59(6): 1036-1049 doi: 10.13343/j.cnki.wsxb.20180353 DU Rui, YU Min, CHENG Jingguang, et al. Diversity and sulfur oxidation characteristics of cultivable sulfur oxidizing bacteria in hydrothermal fields of Okinawa Trough[J]. Acta microbiologica Sinica, 2019, 59(6): 1036-1049. doi: 10.13343/j.cnki.wsxb.20180353
[12] Venter J C, Remington K, Heidelberg J F, et al. Environmental genome shotgun sequencing of the Sargasso Sea[J]. Science, 2004, 304(5667): 66-74. doi: 10.1126/science.1093857
[13] 刘明华, 王健鑫, 俞凯成, 等. 东海陆架表层沉积物细菌群落结构及地理分布研究[J]. 海洋与湖沼, 2015, 46(5): 1119-1131 LIU Minghua, WANG Jianxin, YU Kaicheng, et al. Community structure and geographical distribution of bacterial on surface layer sediments in the east China Sea[J]. Oceanologia et Limnologia Sinica, 2015, 46(5): 1119-1131.
[14] 王苑如, 崔鸿鹏, 李继东, 等. 西太平洋多金属结核区表层沉积物细菌群落结构及其对沉积扰动的响应[J]. 海洋学研究, 2021, 39(2): 21-32 WANG Yuanru, CUI Hongpeng, LI Jidong, et al. The structure of bacterial communities and its response to the sedimentary disturbance in the surface sediment of western Pacific polymetallic nodule area[J]. Journal of Marine Sciences, 2021, 39(2): 21-32.
[15] Inagaki F, Kuypers M M M, Tsunogai U, et al. Microbial community in a sediment-hosted CO2 lake of the southern Okinawa Trough hydrothermal system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(38): 14164-14169.
[16] Yanagawa K, Ijiri A, Breuker A, et al. Defining boundaries for the distribution of microbial communities beneath the sediment-buried, hydrothermally active seafloor[J]. The ISME Journal, 2017, 11(2): 529-542. doi: 10.1038/ismej.2016.119
[17] Nakagawa S, Takai K, Inagaki F, et al. Variability in microbial community and venting chemistry in a sediment-hosted backarc hydrothermal system: impacts of subseafloor phase-separation[J]. FEMS Microbiology Ecology, 2005, 54(1): 141-155. doi: 10.1016/j.femsec.2005.03.007
[18] Wang L, Yu M, Liu Y, et al. Comparative analyses of the bacterial community of hydrothermal deposits and seafloor sediments across Okinawa Trough[J]. Journal of Marine Systems, 2018, 180: 162-172. doi: 10.1016/j.jmarsys.2016.11.012
[19] Yanagawa K, Breuker A, Schippers A, et al. Microbial community stratification controlled by the subseafloor fluid flow and geothermal gradient at the Iheya North hydrothermal field in the Mid-Okinawa Trough (Integrated Ocean Drilling Program Expedition 331)[J]. Applied and Environmental Microbiology, 2014, 80(19): 6126-6135. doi: 10.1128/AEM.01741-14
[20] Yanagawa K, Morono Y, de Beer D, et al. Metabolically active microbial communities in marine sediment under high-CO2 and low-pH extremes[J]. The ISME Journal, 2013, 7(3): 555-567. doi: 10.1038/ismej.2012.124
[21] Zhang J, Sun Q L, Zeng Z G, et al. Microbial diversity in the deep-sea sediments of Iheya North and Iheya Ridge, Okinawa Trough[J]. Microbiological Research, 2015, 177: 43-52. doi: 10.1016/j.micres.2015.05.006
[22] Rona P A, Scott S D. A special issue on sea-floor hydrothermal mineralization: new perspectives: preface[J]. Economic Geology, 1993, 88(8): 1935-1976. doi: 10.2113/gsecongeo.88.8.1935
[23] 邵珂, 陈建平, 任梦依. 印度洋中脊多金属硫化物矿产资源定量预测与评价[J]. 海洋地质与第四纪地质, 2015, 35(5): 125-133 SHAO Ke, CHEN Jianping, REN Mengyi. Quantitative prediction and evaluation of polymetallic sulfide mineral deposits along the central Indian ocean ridge[J]. Marine Geology & Quaternary Geology, 2015, 35(5): 125-133.
[24] Walsh J J. Importance of continental margins in the marine biogeochemical cycling of carbon and nitrogen[J]. Nature, 1991, 350(6313): 53-55. doi: 10.1038/350053a0
[25] 胡思谊. 冲绳海槽南部S3岩心沉积物的矿物学和地球化学研究[D]. 中国科学院大学(中国科学院海洋研究所)博士学位论文, 2020 HU Siyi. Mineralogical and geochemical study of sediment core S3 from the southern Okinawa Trough[D]. Doctor Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2020.
[26] 杨宝菊, 吴永华, 刘季花, 等. 冲绳海槽表层沉积物元素地球化学及其对物源和热液活动的指示[J]. 海洋地质与第四纪地质, 2018, 38(2): 25-37 YANG Baoju, WU Yonghua, LIU Jihua, et al. Elemental geochemistry of surface sediments in Okinawa Trough and its implications for provenance and hydrothermal activity[J]. Marine Geology & Quaternary Geology, 2018, 38(2): 25-37.
[27] 赵维殳, 肖湘. 深海热液区微生物群落与环境适应性机理[J]. 中国科学: 生命科学, 2023, 53(5): 660-671 ZHAO Weishu, XIAO Xiang. Microbiome and environmental adaption mechanisms in deep-sea hydrothermal vents[J]. Scientia Sinica: Vitae, 2023, 53(5): 660-671.
[28] 杨娅敏. 冲绳海槽南部浊流沉积层中的硫化物特征研究[D]. 中国科学院大学(中国科学院海洋研究所)博士学位论文, 2021 YANG Yamin. Study on the characteristics of turbidite sediments hosted-sulfides deposit from the southern Okinawa Trough[D]. Doctor Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2021.
[29] Zeng Z G, Chen S, Ma Y, et al. Chemical compositions of mussels and clams from the Tangyin and Yonaguni Knoll IV hydrothermal fields in the southwestern Okinawa Trough[J]. Ore Geology Reviews, 2017, 87: 172-191. doi: 10.1016/j.oregeorev.2016.09.015
[30] 尚鲁宁, 陈磊, 张训华, 等. 冲绳海槽南部海底热液活动区地形地貌特征及成因分析[J]. 海洋地质与第四纪地质, 2019, 39(4): 12-22 SHANG Luning, CHEN Lei, ZHANG Xunhua, et al. Topographic features of the hydrothermal field and their genetic mechanisms in southern Okinawa Trough[J]. Marine Geology & Quaternary Geology, 2019, 39(4): 12-22.
[31] Atlas R M. Handbook of Microbiological Media[M]. Boca Raton: CRC Press, 1993: 527.
[32] Ruby E G, Wirsen C O, Jannasch H W. Chemolithotrophic sulfur-oxidizing bacteria from the Galapagos rift hydrothermal vents[J]. Applied and Environmental Microbiology, 1981, 42(2): 317-324. doi: 10.1128/aem.42.2.317-324.1981
[33] Das A, Cao W R, Zhang H J, et al. Carbon fixation in sediments of Sino-Pacific seas-differential contributions of bacterial and archaeal domains[J]. Journal of Marine Systems, 2017, 175: 15-23. doi: 10.1016/j.jmarsys.2017.06.005
[34] Cao W R, Lu D C, Sun X K, et al. Seonamhaeicola maritimus sp. nov., isolated from coastal sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(2): 902-908.
[35] Wang F, Men X, Zhang G, et al. Assessment of 16S rRNA gene primers for studying bacterial community structure and function of aging flue-cured tobaccos[J]. AMB Express, 2018, 8(1): 182. doi: 10.1186/s13568-018-0713-1
[36] Tamura K, Stecher G, Kumar S. MEGA11: molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution, 2021, 38(7): 3022-3027. doi: 10.1093/molbev/msab120
[37] Felsenstein J. Evolutionary trees from DNA sequences: a maximum likelihood approach[J]. Journal of Molecular Evolution, 1981, 17(6): 368-376. doi: 10.1007/BF01734359
[38] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. doi: 10.1007/BF01731581
[39] Nossa C W, Oberdorf W E, Yang L Y, et al. Design of 16S rRNA gene primers for 454 pyrosequencing of the human foregut microbiome[J]. World Journal of Gastroenterology, 2010, 16(33): 4135-444. doi: 10.3748/wjg.v16.i33.4135
[40] Yang Y M, Zeng Z G, Yin X B, et al. Mineralogy, geochemistry, and sulfur isotope characteristics of sediment-hosted hydrothermal sulfide minerals from the southern Okinawa Trough[J]. Acta Oceanologica Sinica, 2021, 40(10): 129-143. doi: 10.1007/s13131-021-1836-9
[41] 赵一阳, 翟世奎, 李永植, 等. 冲绳海槽中部热水活动的新记录[J]. 科学通报, 1996, 41(14): 1307-1310 ZHAO Yiyang, ZHAI Shikui, LI Yongzhi, et al. New records of submarine hydrothermal activity in middle part of the Okinawa Trough[J]. Chinese Science Bulletin, 1997, 42(7): 574-577.
[42] 蒋富清, 李安春, 李铁刚. 冲绳海槽北端表层沉积物过渡元素地球化学特征[J]. 海洋与湖沼, 2006, 37(1): 75-83 doi: 10.3321/j.issn:0029-814X.2006.01.012 JIANG Fuqing, LI Anchun, LI Tiegang. Geochemical characteristics of transition elements in surface sediments northern Okinawa Trough[J]. Oceanologia et Limnologia Sinica, 2006, 37(1): 75-83. doi: 10.3321/j.issn:0029-814X.2006.01.012
[43] Edlund A, Hårdeman F, Jansson J K, et al. Active bacterial community structure along vertical redox gradients in Baltic Sea sediment[J]. Environmental Microbiology, 2008, 10(8): 2051-2063. doi: 10.1111/j.1462-2920.2008.01624.x
[44] Chu H Y, Sun H B, Tripathi B M, et al. Bacterial community dissimilarity between the surface and subsurface soils equals horizontal differences over several kilometers in the western Tibetan Plateau[J]. Environmental Microbiology, 2016, 18(5): 1523-1533. doi: 10.1111/1462-2920.13236
[45] Teske A P. Microbial community composition in deep marine subsurface sediments of ODP Leg 201: sequencing surveys and cultivations[M]//Jørgensen B B, D'Hondt S L, Miller D J. Proceedings of the Ocean Drilling Program Scientific Results. 2006.
[46] Pérez-Rodríguez I, Bohnert K A, Cuebas M, et al. Detection and phylogenetic analysis of the membrane-bound nitrate reductase (Nar) in pure cultures and microbial communities from deep-sea hydrothermal vents[J]. FEMS Microbiology Ecology, 2013, 86(2): 256-267. doi: 10.1111/1574-6941.12158
[47] Rajasabapathy R, Mohandass C, Colaco A, et al. Culturable bacterial phylogeny from a shallow water hydrothermal vent of Espalamaca (Faial, Azores) reveals a variety of novel taxa[J]. Current Science, 2014, 106(1): 58-69.
[48] Martins A, Tenreiro T, Andrade G, et al. Photoprotective bioactivity present in a unique marine bacteria collection from Portuguese deep sea hydrothermal vents[J]. Marine Drugs, 2013, 11(5): 1506-1523. doi: 10.3390/md11051506
[49] Kaye J Z, Sylvan J B, Edwards K J, et al. Halomonas and Marinobacter ecotypes from hydrothermal vent, subseafloor and deep-sea environments[J]. FEMS Microbiology Ecology, 2011, 75(1): 123-133. doi: 10.1111/j.1574-6941.2010.00984.x
[50] Cao H L, Wang Y, Lee O O, et al. Microbial sulfur cycle in two hydrothermal chimneys on the southwest Indian Ridge[J]. mBio, 2014, 5(1): e00980-13.
[51] Mohandass C, Rajasabapathy R, Ravindran C, et al. Bacterial diversity and their adaptations in the shallow water hydrothermal vent at D. João de Castro Seamount (DJCS), Azores, Portugal[J]. Cahiers de Biologie Marine, 2012, 53(1): 65-76.
[52] Dick G J, Lee Y E, Tebo B M. Manganese(II)-oxidizing Bacillus spores in Guaymas basin hydrothermal sediments and plumes[J]. Applied and Environmental Microbiology, 2006, 72(5): 3184-3190. doi: 10.1128/AEM.72.5.3184-3190.2006
[53] Liao L, Xu X W, Jiang X W, et al. Microbial diversity in deep-sea sediment from the cobalt-rich crust deposit region in the Pacific Ocean[J]. FEMS Microbiology Ecology, 2011, 78(3): 565-585. doi: 10.1111/j.1574-6941.2011.01186.x
[54] Xu M X, Wang F P, Meng J, et al. Construction and preliminary analysis of a metagenomic library from a deep-sea sediment of East Pacific Nodule province[J]. FEMS Microbiology Ecology, 2007, 62(3): 233-241. doi: 10.1111/j.1574-6941.2007.00377.x
[55] 魏曼曼. 劳盆地深海热液喷口沉积物中微生物群落结构研究[D]. 中南大学硕士学位论文, 2010 WEI Manman. Study on microbial community structures in sediments from Lau Basin deep-sea hydrothermal vents[D]. Master Dissertation of Central South University, 2010.
[56] Flores G E, Campbell J H, Kirshtein J D, et al. Microbial community structure of hydrothermal deposits from geochemically different vent fields along the Mid-Atlantic Ridge[J]. Environmental Microbiology, 2011, 13(8): 2158-2171. doi: 10.1111/j.1462-2920.2011.02463.x
[57] Sylvan J B, Toner B M, Edwards K J. Life and death of deep-sea vents: bacterial diversity and ecosystem succession on inactive hydrothermal sulfides[J]. mBio, 2012, 3(1): e00279-11.
[58] Cao W R, Wang L S, Saren G, et al. Variable microbial communities in the non-hydrothermal sediments of the Mid-Okinawa Trough[J]. Geomicrobiology Journal, 2020, 37(8): 774-782. doi: 10.1080/01490451.2020.1776800
[59] 张伟. 热带西太平洋深海沉积物微生物多样性及群落结构特征研究[D]. 中国科学院研究生院(海洋研究所)硕士学位论文, 2010 ZHANG Wei. Microbial diversity and community structure in marine sediments from the tropical western Pacific[D]. Master Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2010.
[60] Lee S Y, Huh C A, Su C C, et al. Sedimentation in the Southern Okinawa Trough: enhanced particle scavenging and teleconnection between the Equatorial Pacific and western Pacific margins[J]. Deep Sea Research Part I: Oceanographic Research Papers, 2004, 51(11): 1769-1780. doi: 10.1016/j.dsr.2004.07.008
[61] 邵宗泽. 深海热液区化能自养微生物多样性、代谢特征与环境作用[J]. 科学通报, 2018, 63(36): 3902-3910 doi: 10.1360/N972018-00980 SHAO Zongze. Diversity and metabolism of prokaryotic chemoautotrophs and their interactions with deep-sea hydrothermal environments[J]. Chinese Science Bulletin, 2018, 63(36): 3902-3910. doi: 10.1360/N972018-00980
[62] Li Y X, Li F C, Zhang X W, et al. Vertical distribution of bacterial and archaeal communities along discrete layers of a deep-sea cold sediment sample at the East Pacific Rise (~13°N)[J]. Extremophiles, 2008, 12(4): 573-585. doi: 10.1007/s00792-008-0159-5
[63] Wang P, Xiao X, Wang F P. Phylogenetic analysis of Archaea in the deep-sea sediments of west Pacific Warm Pool[J]. Extremophiles, 2005, 9(3): 209-217. doi: 10.1007/s00792-005-0436-5




 下载:
下载: